Badanie ujawniło mechanizm chroniący tkankę po nieprawidłowej ekspresji genów
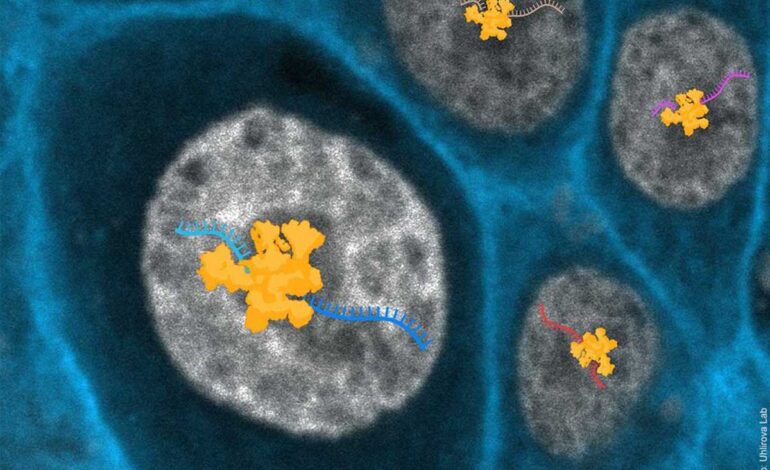
Materiał genetyczny, występujący w postaci DNA, zawiera informacje istotne dla prawidłowego funkcjonowania komórek ludzkich i zwierzęcych. Informacje te generują RNA, cząsteczkę pośrednią między DNA a białkami, budulcem komórek. Podczas tego procesu informacje genetyczne są dostosowywane w sposób dostosowany do potrzeb każdej komórki. Niepotrzebne informacje (introny) są usuwane z RNA, podczas gdy podstawowe składniki białek (eksony) zostają zachowane.
Zespół naukowców kierowany przez profesor dr Mirkę Uhlirovą z klastra doskonałości CECAD w badaniach nad starzeniem się na Uniwersytecie w Kolonii odkrył, że gdy przetwarzanie informacji przebiega nieprawidłowo, aktywowany jest kompleks białkowy (heterodimer C/EBP). Kompleks ten wprowadza komórkę w stan uśpienia znany jako starzenie się komórkowe.
W organizmach posiadających jądro komórkowe (eukarionty) występuje maszyna molekularna zwana spliceosomem. Spliceosom przeprowadza „splatanie” – proces, który usuwa niepotrzebne sekcje (introny) i łączy istotne części (egzony) w celu utworzenia informacyjnego RNA (mRNA). Dysfunkcje spliceosomu mogą powodować choroby zwane spliceosomopatiami, wpływające na różne tkanki i powodujące takie stany, jak zwyrodnienie siatkówki lub zespół mielodysplastyczny wpływający na szpik kostny.
Wykorzystując muszkę owocową Drosophila melanogaster jako organizm modelowy, naukowcy z laboratorium Uhlirova zbadali, w jaki sposób komórki reagują na nieprawidłowe działanie spliceosomów podczas rozwoju. W badaniu wykorzystano genomikę i genetykę funkcjonalną, aby zrozumieć rolę poszczególnych genów i ich interakcje.
Badania wykazały, że komórki z wadliwym spliceosomalnym U5 snRNP aktywują reakcję sygnalizacyjną stresu i wykazują zachowania komórkowe charakterystyczne dla starzenia się komórek. Starzenie się komórek zmienia ważne funkcje komórek, zapobiegając podziałom komórek i jednocześnie promując wydzielanie. Starzenie się jest reakcją ochronną mającą na celu zachowanie uszkodzonych komórek, ponieważ ich natychmiastowa eliminacja może być bardziej szkodliwa. Jednakże nagromadzenie starzejących się komórek może mieć negatywny wpływ na tkanki i cały organizm, powodując konieczność ich ostatecznego usunięcia.
Zespół zidentyfikował kompleks białkowy zwany heterodimerem C/EBP, w szczególności Xrp1/Irbp18, jako kluczowy czynnik napędzający reakcję na stres spowodowaną nieprawidłowym splicingiem. Zwiększone poziomy Xrp1/Irbp18 w uszkodzonych komórkach doprowadziły do zwiększonej produkcji białek i wywołały stan przypominający starzenie się komórek.
Profesor dr Mirka Uhlirova z klastra doskonałości CECAD w badaniach nad starzeniem się na Uniwersytecie w Kolonii powiedziała: „Starzenie się to miecz obosieczny. Jedną z zalet starzejących się komórek jest to, że nie wszystkie są eliminowane jednocześnie przez śmierć komórki, co pozwala zachować integralność tkanki. Przecież częściowo nienaruszona tkanka jest lepsza niż żadna. Jednak komórki te stwarzają problemy w dłuższej perspektywie, ponieważ ich akumulacja sprzyja chorobom i starzeniu się”.
„Działający spliceosom jest podstawowym warunkiem zdrowych komórek, tkanek i całego organizmu. Dodatkowe badanie zidentyfikowanego przez nas programu sygnalizacji stresu będzie ważne, aby dokładniej rozwikłać złożone reakcje wywoływane przez defekty w podstawowych maszynach kontrolujących ekspresję genów – i tego, jak możemy na nie wpływać”.
W przyszłości wyniki mogą przyczynić się do opracowania podejść terapeutycznych w leczeniu chorób spowodowanych nieprawidłowym działaniem spliceosomu”.
Numer czasopisma:
- Mirka Uhlirova i wsp.: „Xrp1 reguluje program reakcji na stres na dysfunkcję spliceosomu”, Nucleic Acids Research, 2 lutego 2024 r. DOI: 10.1093/nar/gkae055






