Naukowcy odkrywają system nowej generacji do programowalnego projektowania genomu
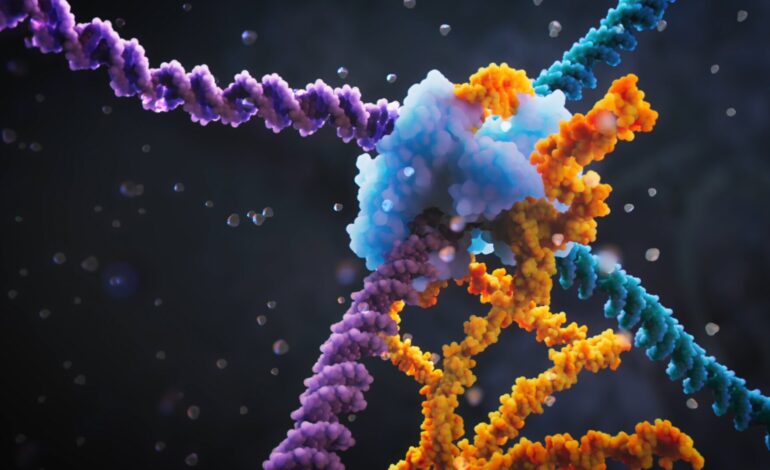
Wizualizacja mechanizmu rekombinazy mostkowej. Źródło: Nauki wizualne
× zamknąć
Wizualizacja mechanizmu rekombinazy mostkowej. Źródło: Nauki wizualne
Dokonując kroku naprzód w inżynierii genetycznej, zespół naukowców z Instytutu Arc odkrył mechanizm rekombinazy mostkowej, precyzyjne i potężne narzędzie do rekombinacji i zmiany układu DNA w programowalny sposób.
Badanie opublikowane dzisiaj w Natura donoszą o odkryciu pierwszej rekombinazy DNA, która wykorzystuje niekodujący RNA do selekcji specyficznej dla sekwencji cząsteczek DNA docelowego i dawcy. Ten mostkowy RNA jest programowalny, co pozwala użytkownikowi określić dowolną pożądaną genomową sekwencję docelową i dowolną cząsteczkę DNA dawcy, która ma zostać wstawiona.
„System mostkowego RNA to zasadniczo nowy mechanizm programowania biologicznego” – powiedział Hsu, starszy autor badania i główny badacz Arc Institute oraz adiunkt bioinżynierii na Uniwersytecie Kalifornijskim w Berkeley. „Rekombinacja mostkowa może uniwersalnie modyfikować materiał genetyczny poprzez insercję, wycięcie, inwersję i inne specyficzne dla sekwencji, umożliwiając procesor tekstu dla żywego genomu poza CRISPR”.
Głównymi autorami odkrycia byli starszy naukowiec Arc Matthew Durrant i absolwent bioinżynierii Uniwersytetu Kalifornijskiego w Berkeley Nick Perry. Badania opracowano we współpracy z laboratoriami Silvany Konermann, głównego badacza Arc Institute i adiunkta biochemii na Uniwersytecie Stanforda oraz Hiroshiego Nishimasu, profesora biologii strukturalnej na Uniwersytecie Tokijskim.
Programowalne RNA
System rekombinacji mostkowej wywodzi się z elementów o sekwencji insercyjnej 110 (IS110), jednego z niezliczonych typów elementów ulegających transpozycji – czyli „skaczących genów” – które wycinają się i wklejają, aby przemieszczać się w obrębie genomów drobnoustrojów i pomiędzy nimi. Elementy transpozycyjne występują we wszystkich formach życia i ewoluowały w profesjonalne maszyny do manipulacji DNA, aby przetrwać. Elementy IS110 są bardzo minimalne i składają się jedynie z genu kodującego enzym rekombinazę oraz flankujących segmentów DNA, które do tej pory pozostawały tajemnicą.
Laboratorium Hsu odkryło, że kiedy IS110 wycina się z genomu, niekodujące końce DNA łączą się ze sobą, tworząc cząsteczkę RNA – mostkowy RNA – który składa się w dwie pętle. Jedna pętla wiąże się z samym elementem IS110, podczas gdy druga pętla wiąże się z docelowym DNA, do którego zostanie wstawiony element. Mostkowy RNA jest pierwszym przykładem bispecyficznej cząsteczki kierującej, określającej sekwencję zarówno DNA docelowego, jak i dawcy poprzez interakcje parowania zasad.
Patrick Hsu, Nick Perry i Matt Durrant omawiają nowo odkryty mechanizm rekombinazy mostkowej. Źródło: Ray Rudolph
× zamknąć
Patrick Hsu, Nick Perry i Matt Durrant omawiają nowo odkryty mechanizm rekombinazy mostkowej. Źródło: Ray Rudolph
Każda pętla mostkowego RNA jest programowalna niezależnie, co pozwala naukowcom na mieszanie i dopasowywanie dowolnych sekwencji DNA docelowego i dawcy będących przedmiotem zainteresowania. Oznacza to, że system może wyjść daleko poza swoją naturalną rolę, polegającą na wstawianiu samego elementu IS110, zamiast tego umożliwiając wstawienie dowolnego pożądanego ładunku genetycznego – na przykład funkcjonalnej kopii wadliwego genu powodującego chorobę – w dowolne miejsce genomu. W tej pracy zespół wykazał ponad 60% skuteczność insercji pożądanego genu w E. coli z ponad 94% swoistością w zakresie prawidłowej lokalizacji genomowej.
„Te programowalne mostkowe RNA odróżniają IS110 od innych znanych rekombinaz, którym brakuje składnika RNA i których nie można zaprogramować” – powiedział doktorant Nick Perry. „To tak, jakby mostek RNA był uniwersalnym zasilaczem, dzięki któremu IS110 będzie kompatybilny z każdym gniazdkiem.”
Wizualizacja mechanizmu rekombinazy mostkowej podkreślająca DNA transpozonu i miejsce docelowe genomu. Źródło: Nauki wizualne
× zamknąć
Wizualizacja mechanizmu rekombinazy mostkowej podkreślająca DNA transpozonu i miejsce docelowe genomu. Źródło: Nauki wizualne
Uzupełnieniem odkrycia laboratorium Hsu jest współpraca z laboratorium dr Hiroshi Nishimasu na Uniwersytecie Tokijskim, opublikowana także dzisiaj w Natura. Laboratorium Nishimasu wykorzystało kriomikroskopię elektronową do określenia struktur molekularnych kompleksu RNA z mostkiem rekombinazy związanego z DNA docelowym i dawcy, przechodząc kolejno przez kluczowe etapy procesu rekombinacji.
Dzięki dalszym badaniom i rozwojowi mechanizm mostkowy może zapoczątkować trzecią generację systemów kierowanych przez RNA, wykraczającą poza mechanizmy cięcia DNA i RNA w ramach CRISPR i interferencji RNA (RNAi), oferując ujednolicony mechanizm programowalnych rearanżacji DNA. Rekombinaza mostkowa, kluczowa dla dalszego rozwoju systemu rekombinacji mostkowej w projektowaniu genomu ssaków, łączy obie nici DNA bez uwalniania wyciętych fragmentów DNA, omijając kluczowe ograniczenia obecnych, najnowocześniejszych technologii edycji genomu.
Wizualizacja mechanizmu rekombinazy mostkowej z uwydatnieniem pętli wiązania donora i celu. Źródło: Nauki wizualne
× zamknąć
Wizualizacja mechanizmu rekombinazy mostkowej z uwydatnieniem pętli wiązania donora i celu. Źródło: Nauki wizualne
„Mechanizm rekombinacji pomostowej rozwiązuje niektóre z najbardziej podstawowych wyzwań stojących przed innymi metodami edycji genomu” – powiedział współkierownik badań Durrant. „Możliwość programowej zmiany układu dowolnych dwóch cząsteczek DNA otwiera drzwi do przełomów w projektowaniu genomu”.
Inni współautorzy to James Pai i Aditya Jangid (Instytut Arc i Uniwersytet Kalifornijski w Berkeley); Januka Athukoralage, John McSpedon i April Pawluk (Instytut Arc); i Masahiro Hiraizumi (Uniwersytet Tokijski).
Więcej informacji:
Patrick Hsu, Bridge RNAs bezpośrednia programowalna rekombinacja DNA docelowego i dawcy, Natura (2024). DOI: 10.1038/s41586-024-07552-4. www.nature.com/articles/s41586-024-07552-4
Hiraizumi, M i in. Strukturalny mechanizm rekombinacji kierowanej przez RNA mostkowy, Natura (2024). DOI: 10.1038/s41586-024-07570-2 www.nature.com/articles/s41586-024-07570-2
Informacje o czasopiśmie:
Natura
Dostarczone przez Instytut Arc






