Naukowcy wyjaśniają strukturę przestrzenną i mechanizmy molekularne „głównego redaktora”, nowatorskiego narzędzia do edycji genów
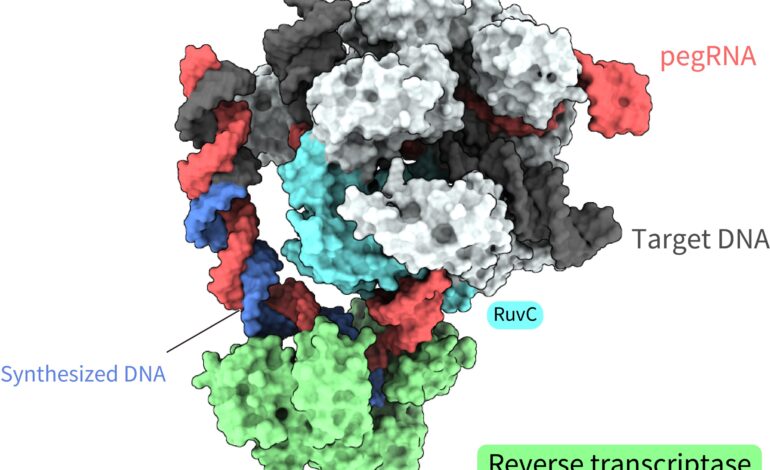
Model powierzchniowy kompleksu SpCas9 – odwrotna transkryptaza – pegRNA – docelowy DNA. Główny edytor, składający się ze SpCas9 i odwrotnej transkryptazy, dokonuje odwrotnej transkrypcji sekwencji matrycy w pegRNA, co skutkuje włączeniem pożądanych zmian do docelowego miejsca w genomie. Heterodupleks RNA – DNA tworzy się wzdłuż domeny nukleazy RuvC SpCas9. Źródło: Shuto i in. 2024
× zamknąć
Model powierzchniowy kompleksu SpCas9 – odwrotna transkryptaza – pegRNA – docelowy DNA. Główny edytor, składający się ze SpCas9 i odwrotnej transkryptazy, dokonuje odwrotnej transkrypcji sekwencji matrycy w pegRNA, co skutkuje włączeniem pożądanych zmian do docelowego miejsca w genomie. Heterodupleks RNA – DNA tworzy się wzdłuż domeny nukleazy RuvC SpCas9. Źródło: Shuto i in. 2024
Wspólne badania prowadzone przez Yutaro Shuto, Ryoyę Nakagawę i Osamu Nureki z Uniwersytetu Tokijskiego określiły strukturę przestrzenną różnych procesów nowatorskiego narzędzia do edycji genów zwanego „głównym edytorem”. Analiza funkcjonalna oparta na tych strukturach ujawniła również, w jaki sposób „główny redaktor” może osiągnąć odwrotną transkrypcję, syntetyzując DNA z RNA, bez „przecinania” obu nici podwójnej helisy.
Wyjaśnienie tych mechanizmów molekularnych w znacznym stopniu przyczynia się do zaprojektowania narzędzi do edycji genów, wystarczająco dokładnych na potrzeby terapii genowych. Wyniki publikuje się w czasopiśmie Natura.
Nagrodę Nobla w dziedzinie chemii 2020 przyznano Jennifer Doudna i Emmanuelle Charpentier za opracowanie przełomowej, a jednocześnie prostej metody edycji DNA, czyli „planu” żywych organizmów. Chociaż ich odkrycie otworzyło nowe możliwości badań, dokładność metody i obawy dotyczące bezpieczeństwa związane z „przecięciem” obu nici DNA ograniczyły jej zastosowanie w terapii genowej. W związku z tym trwają badania mające na celu opracowanie narzędzi pozbawionych tych wad.
Jednym z takich narzędzi jest główny system edycji, kompleks cząsteczek składający się z dwóch składników. Jednym ze składników jest główny edytor, który łączy w sobie białko SpCas9 stosowane w pierwszej technologii edycji genów CRISPR-Cas oraz odwrotną transkryptazę, enzym dokonujący transkrypcji RNA na DNA.
Drugim składnikiem jest główny przewodnik RNA (pegRNA), zmodyfikowany przewodnik RNA, który identyfikuje docelową sekwencję w DNA i koduje pożądaną edycję. W tym kompleksie główny redaktor działa jak „edytor tekstu”, dokładnie zastępując informacje genomiczne. Narzędzie zostało już z powodzeniem wdrożone w żywych komórkach organizmów, takich jak rośliny, danio pręgowany i myszy. Jednakże dokładny sposób, w jaki ten kompleks molekularny wykonuje każdy etap procesu edycji, nie jest jasny, głównie z powodu braku informacji na temat jego struktury przestrzennej.
„Zaciekawiło nas, jak nienaturalna kombinacja białek Cas9 i odwrotnej transkryptazy współdziała ze sobą” – mówi Shuto, pierwszy autor artykułu.
Strukturalny model krokowy głównego edytora. Zasady pegRNA łączą się w pary z naciętą nicią docelowego DNA, tworząc heterodupleks RNA–DNA na powierzchni domeny nukleazy RuvC (tworzenie RNA–DNA). Odwrotna transkryptaza rozpoznaje heterodupleks RNA–DNA i inicjuje odwrotną transkrypcję (inicjację). Odwrotna transkryptaza konsekwentnie angażuje się w odwrotną transkrypcję sekwencji matrycy wokół miejsca inicjacji, a wydłużony heterodupleks RNA–DNA gromadzi się wzdłuż podłużnej powierzchni SpCas9, czemu towarzyszy rearanżacja docelowego dupleksu DNA (wydłużenie). Odwrotna transkryptaza wykonuje nadmierną odwrotną transkrypcję poza matrycą i kończy odwrotną transkrypcję z powodu kolizji z Cas9, co prowadzi do niepożądanych insercji (terminacja). Źródło: Shuto i in. 2024
× zamknąć
Strukturalny model krokowy głównego edytora. Zasady pegRNA łączą się w pary z naciętą nicią docelowego DNA, tworząc heterodupleks RNA–DNA na powierzchni domeny nukleazy RuvC (tworzenie RNA–DNA). Odwrotna transkryptaza rozpoznaje heterodupleks RNA–DNA i inicjuje odwrotną transkrypcję (inicjację). Odwrotna transkryptaza konsekwentnie angażuje się w odwrotną transkrypcję sekwencji matrycy wokół miejsca inicjacji, a wydłużony heterodupleks RNA–DNA gromadzi się wzdłuż podłużnej powierzchni SpCas9, czemu towarzyszy rearanżacja docelowego dupleksu DNA (wydłużenie). Odwrotna transkryptaza wykonuje nadmierną odwrotną transkrypcję poza matrycą i kończy odwrotną transkrypcję z powodu kolizji z Cas9, co prowadzi do niepożądanych insercji (terminacja). Źródło: Shuto i in. 2024
Zespół badawczy zastosował kriogeniczną mikroskopię elektronową – technikę obrazowania umożliwiającą obserwacje w skali bliskiej atomowej. Metoda wymagała umieszczania próbek w szklistym lodzie, aby chronić je przed potencjalnym uszkodzeniem przez wiązki elektronów, co stwarzało dodatkowe wyzwania.
„Odkryliśmy, że kompleks głównego redaktora jest niestabilny w warunkach eksperymentalnych” – wyjaśnia Shuto. „Tak więc optymalizacja warunków zapewniających stabilność kompleksu była bardzo trudna. Przez długi czas mogliśmy jedynie określić strukturę Cas9”.
Wreszcie, przezwyciężając te wyzwania, naukowcom udało się określić trójwymiarową strukturę kompleksu głównego redaktora w wielu stanach podczas odwrotnej transkrypcji na docelowym DNA.
Struktury ujawniły, że odwrotna transkryptaza wiązała się z kompleksem RNA–DNA, który utworzył się wzdłuż „części” białka Cas9 związanej z rozszczepieniem DNA, czyli rozszczepieniem pojedynczej nici podwójnej helisy. Podczas przeprowadzania odwrotnej transkrypcji odwrotna transkryptaza utrzymywała swoją pozycję względem białka Cas9. Analizy strukturalne i biochemiczne wykazały również, że odwrotna transkryptaza może prowadzić do dodatkowych, niepożądanych insercji.
Odkrycia te otworzyły nowe możliwości zarówno w badaniach podstawowych, jak i stosowanych. Zatem Shuto przedstawia kolejne kroki.
„Naszą strategię określania struktury zastosowaną w tym badaniu można również zastosować do głównych redaktorów składających się z innego białka Cas9 i odwrotnej transkryptazy. Chcemy wykorzystać nowo uzyskane informacje strukturalne, aby doprowadzić do opracowania ulepszonych głównych redaktorów”.
Więcej informacji:
Strukturalne podstawy odwrotnej transkrypcji kierowanej pegRNA przez głównego redaktora, Natura (2024). DOI: 10.1038/s41586-024-07497-810.1
Informacje o czasopiśmie:
Natura