Odkrywanie potencjalnych celów leków w leczeniu nadczynności przytarczyc poprzez wiedzę genetyczną za pomocą analiz randomizacji mendlowskiej i kolokalizacji
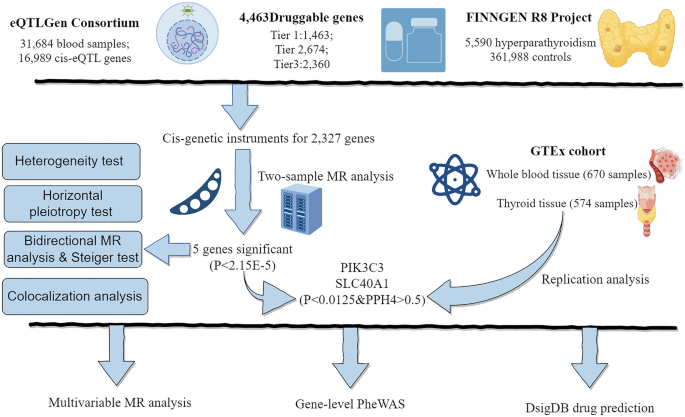
Projekt badania i źródła danych
W niniejszym badaniu starano się zidentyfikować nowe cele terapeutyczne istotne w leczeniu nadczynności przytarczyc (HPT). Potok metodologiczny przedstawiono za pomocą schematu blokowego (ryc. 1) i ukierunkowanego wykresu acyklicznego (ryc. 2), ze szczegółowymi informacjami o źródłach danych podanymi w tabeli uzupełniającej 1. Najpierw przeprowadzono randomizację mendlowską dla dwóch próbek przy użyciu danych eQTL z eQTLGen i nadczynność przytarczyc GWAS z FinnGen w celu wskazania potencjalnych genów sprawczych. Po drugie, ustalenia poddano dalszej walidacji za pomocą testów heterogeniczności, oceny poziomej plejotropii i wykrywania odwrotnej przyczynowości. Po trzecie, analizę powtórzono w danych GTEx w ramach zewnętrznej walidacji. Następnie przeprowadzono wielowymiarową analizę MR w celu określenia łącznych skutków wielu czynników ryzyka. Na koniec zastosowano analizę kolokalizacji bayesowskiej, badania PheWAS i przewidywanie leków, aby zbadać wykonalność zidentyfikowanych celów leków w potencjalnych przyszłych zastosowaniach klinicznych.
Omówienie projektu badania (autor: Figdraw).
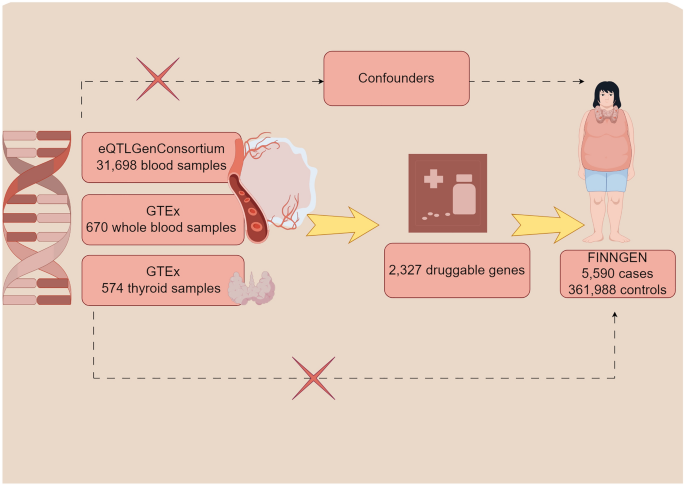
Skierowany wykres acykliczny (DAG) tej pracy (by Figdraw).
Nasze badanie było wtórną analizą publicznie dostępnych danych. W szczególności dane na poziomie podsumowania uzyskano z trzech wielkoskalowych zasobów asocjacyjnych obejmujących cały genom: eQTLGenConsortium (https://eqtlgen.org/)16zbiór danych GTEx (https://gtexportal.org/home/eqtlDashboardPage)17i FinnGen (https://www.finngen.fi/en)18. Włączone badania uzyskały aprobatę etyczną od odpowiednich instytucjonalnych komisji odwoławczych. Niniejsze badanie przeprowadzono zgodnie z obowiązującymi wytycznymi, a listę kontrolną STROBE-MR załączono jako materiał uzupełniający19.
Możliwe do podjęcia działania cele narkotykowe
Do analizy zidentyfikowano łącznie 4464 genów nadających się do stosowania leku, zlokalizowanych na chromosomach autosomalnych z adnotacjami HGNC. Zestaw ten obejmował 1425 genów kodujących docelowe białka będące w bieżącym rozwoju klinicznym, 674 geny powiązane z białkami zaangażowanymi w zatwierdzone leki lub związki oraz 2360 genów należących do ustalonych rodzin docelowych leków. Badanie przeprowadzone przez Chrisa i wsp. dostarcza dalszych szczegółowych informacji na temat tych możliwych celów farmakologicznych20.
Dane eQTL do identyfikacji genetycznych zmiennych instrumentalnych
Genetyczne zmienne instrumentalne uzyskano z dwóch odrębnych zbiorów danych eQTL. Zbiór danych kohorty odkrywczej wykorzystany w tym celu obejmował dane eQTL pochodzące od konsorcjum eQTLGen. Ponadto zestaw danych kohorty replikacji uzyskano z GTEx, zapewniając solidną walidację naszych ustaleń w niezależnych zbiorach danych.
konsorcjum eQTLGen
Zidentyfikowano istotne cis-eQTL dla całego genomu (wskaźnik fałszywych odkryć <0,05) w obrębie ± 1 Mb lokalizacji sondy. Zbiór danych wykorzystany do tej analizy, eQTLGen, obejmuje 16 987 genów i został uzyskany z analizy 31 684 próbek krwi, pobranych głównie od osób pochodzenia europejskiego i charakteryzujących się ogólnie dobrym stanem zdrowia. Podkreślając znaczenie eQTL w badaniach nad opracowywaniem leków ze względu na ich bliskość do genów docelowych i bezpośredni wpływ na ekspresję genów, zawęziliśmy nasz wybór do SNP znajdujących się w odległości 100 kb powyżej miejsc startu transkrypcji lub 100 kb poniżej miejsc końcowych transkrypcji leków nadających się do stosowania geny. W rezultacie zidentyfikowaliśmy eQTL powiązane z 2327 genami podatnymi na leki.
GTEx
Jako niezależne kohorty replikacyjne, dane eQTL dla 670 krwi pełnej 574 i tkanki tarczycy uzyskano z wersji 8 bazy danych GTEx (https://gtexportal.org/home/)21.
Dane wynikowe
Nadczynność przytarczyc
Dane GWAS dotyczące nadczynności przytarczyc pochodzą z FinnGen Release 8 (opublikowanego w grudniu 2022 r. Nadczynność przytarczyc w FinnGen zdefiniowano na podstawie Międzynarodowej Klasyfikacji Chorób (ICD) obejmującej 5590 przypadków (ICD-8(2520), ICD-9( 2520), ICD-10(550)) i 361 988 kontroli.
Czynniki ryzyka
Zidentyfikowaliśmy 12 czynników ryzyka nadczynności przytarczyc obejmujących przewlekłą chorobę nerek22przewlekłe cewkowo-śródmiąższowe zapalenie nerek18Kłębuszkowe zapalenie nerek18Nefropatia IgA18kamica nerkowa18poziom wapnia we krwi23magnez w surowicy/osoczu24surowica/osocze 25-hydroksywitamina D2/D324stężenie fosforanów w surowicy25hormon przytarczyc26zespół złego wchłaniania jelitowego18i osteomalację18 (Tabela uzupełniająca S2).
Mendlowska analiza randomizacji
Do wykonania analizy MR dwóch próbek wykorzystano pakiet TwoSampleMR R (wersja 0.5.7, TwoSampleMR/)27. Przed badaniem MR wdrożono rygorystyczną kontrolę jakości instrumentów SNP. Po pierwsze, warianty o słabej sile instrumentu (statystyka F < 10, gdzie F = (beta/se)2) zostały wykluczone. Warunkowo niezależne warianty w nierównowadze o niskim sprzężeniu (LD r2Następnie wybrano < 0,1 na 1000 genomów w europejskim panelu). Na koniec filtrowanie Steigera usunęło geny, w których SNP wyjaśniały większy wynik niż wariancja ekspozycji (tabela uzupełniająca S3).
W analizie pierwotnej zastosowano współczynnik Walda dla proponowanych instrumentów zawierających pojedynczy SNP. W przypadku proponowanych instrumentów obejmujących więcej niż jeden SNP przyjęto podejście kompleksowe. Obejmowało to zastosowanie metody ważonej odwrotną wariancją (IVW), MR-Eggera i ważonej mediany MR. Metoda IVW zakłada, że wszystkie instrumenty genetyczne są prawidłowe i ma największą moc statystyczną, gdy to założenie jest spełnione 28. Podejście oparte na medianie ważonej pozwala na użycie niektórych (50%) nieprawidłowych instrumentów poprzez ważenie szacunków MR specyficznych dla SNP w oparciu o wielkość i przyjęcie szacunków mediany z błędami standardowymi metodą bootstrap29. MR-Egger pozwala na plejotropię poziomą, w której niektóre SNP wpływają na wynik alternatywnymi ścieżkami, ale ma niższą moc statystyczną. Test przechwytywania MR-Eggera pozwala określić obecność plejotropii poziomej30. Aby uwzględnić wielokrotne testowanie w analizach wrażliwości, wprowadzono poprawki Bonferroniego w celu ustalenia skorygowanych progów istotności. W kohorcie eQTLGen P wartości poniżej 2,15e−05 (obliczone wg P= 0,05/2327, 2327 to liczba genów nadających się do stosowania leku w danych eQTLGen) uznano za istotne. Następnie do genów zidentyfikowanych jako istotne zastosowano procedury kontroli jakości, zapewniając spójność w kierunku szacowanych efektów w przypadku trzech metod i potwierdzając brak plejotropii poziomej za pomocą testu MR-Eggera. Geny przechodzące kontrolę jakości poddano dalszej ocenie w kohorcie GTEx. Konserwatywny próg istotności wynoszący PZastosowano < 1,25e-02 (obliczone jako 0,05/4 dla poprawki Bonferroniego, 4 to liczba genów replikowanych w danych GTEx). Replikacja przy tym rygorystycznym punkcie odcięcia zwiększyła niezawodność powiązań, zmniejszając prawdopodobieństwo fałszywych alarmów, zapewniając zewnętrzną weryfikację uzupełniającą oryginalną analizę wykrywania eQTLGen.
Dodatkowo przeprowadzono wielowymiarową analizę MR przy użyciu pakietu MVMR R, badając powiązania PIK3C3 i SLC40A1 z ryzykiem nadczynności przytarczyc, po uwzględnieniu drugiej zmiennej. Jako rozszerzenie jednowymiarowej MR, wieloczynnikowa MR może wyznaczyć łączne skutki przyczynowe wielu czynników ryzyka 31. W przypadku PIK3C3 skorygowano wpływ złego wchłaniania jelitowego18. W przypadku SLC40A1 wprowadzono korekty ze względu na potencjalne skutki przewlekłej choroby nerek22poziom wapnia we krwi23i kłębuszkowe zapalenie nerek18.
Wykrywanie odwrotnej przyczynowości
Stosując się do analogicznych kryteriów przesiewowych stosowanych w przypadku eQTL, ze zbioru danych GWAS firmy FinnGen skrupulatnie wybrano instrumenty genetyczne do wykrywania nadczynności przytarczyc. Instrumenty te wykorzystano następnie w dwukierunkowej analizie MR w celu ustalenia możliwej odwrotnej przyczyny. Oceny efektów uzyskano przy użyciu trzech różnych metod: MR-odwrotnej wariancji ważonej (MR-IVW), MR-Eggera i ważonej mediany. Ponadto przeprowadzono procedurę filtrowania Steigera, aby potwierdzić kierunkowość związku między ekspresją loci cech ilościowych (eQTL) a nadczynnością przytarczyc32. Istotność statystyczną ustalono na progu P< 0,05, co stanowi rygorystyczne kryterium ważności i wiarygodności obserwowanych powiązań.
Analiza kolokalizacji
W przypadku genów ze znaczącymi powiązaniami randomizacji mendlowskiej zarówno w kohortach eQTLGen, jak i GTEx, analizę kolokalizacji przeprowadzono przy użyciu pakietu coloc R z domyślnymi priorytetami 33. W tym podejściu bayesowskim sprawdzano, czy powiązania między ekspresją genów a nadczynnością przytarczyc wynikają ze wspólnych wariantów przyczynowych w danym locus, a nie z braku równowagi powiązań. W szczególności oceniono pięć wzajemnie wykluczających się hipotez: (H0) brak związku z którąkolwiek cechą; (H1) skojarzenie tylko z wyrażeniem; (H2) tylko związek z chorobą; (H3) związek z obiema cechami, ale niezależnymi wariantami przyczynowymi; (H4) związek z obiema cechami wynikający ze wspólnego wariantu przyczynowego34. Dla każdej hipotezy określono prawdopodobieństwa późniejsze. Prawdopodobieństwa wcześniejsze ustalono na 1e-04 tylko dla cechy 1 (p1) i tylko cechy 2 (p2) oraz 1e-05 dla obu cech (p12). Mocne dowody kolokalizacji zdefiniowano jako prawdopodobieństwo późniejsze ≥ 0,8 dla H4, przy wartościach od 0,5 do 0,8 sugerujących umiarkowaną kolokalizację.
Analiza asocjacji obejmująca cały fenomen
Wykorzystując portal AstraZeneca PheWAS (i bazę danych PheWeb (PheWAS przeprowadzono w celu dokładnej oceny poziomej plejotropii możliwych celów terapeutycznych i prawdopodobnych działań niepożądanych)35,36.
Przewidywanie kandydatów na leki
W trakcie tego badania zidentyfikowane geny docelowe są przesyłane do bazy danych sygnatur leków (DSigDB, w celu oceny interakcji białko-lek. DSigDB stanowi istotne repozytorium, w którym znajduje się 22 527 zestawów genów i 17 389 różnych związków powiązanych z 19 531 genami). Ta wszechstronna baza danych ułatwia ustalenie powiązań między środkami farmaceutycznymi, różnymi substancjami chemicznymi i ich odpowiednimi genami docelowymi. PIK3C3 i SLC40A1 analizowano przy użyciu bazy danych leków DSigDB w witrynie Enrichr (w celu zidentyfikowania potencjalnych czynników docelowych37.






