Odszyfrowanie różnorodności genetycznej Malbeca w celu uzyskania doskonałości klonalnej
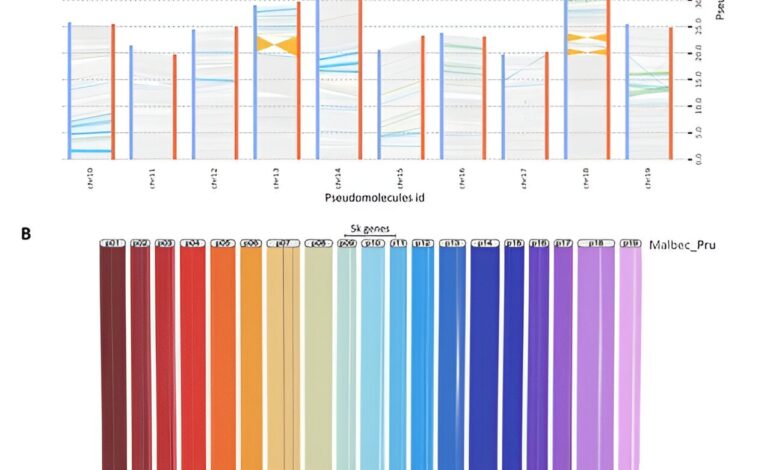
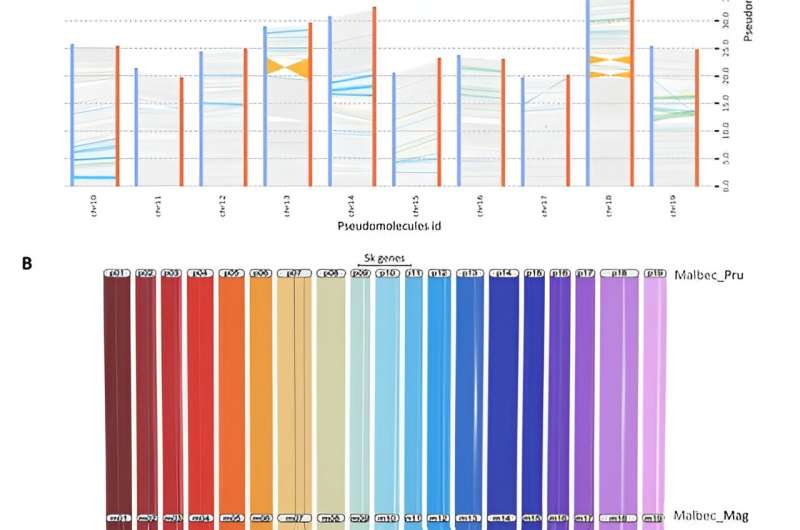
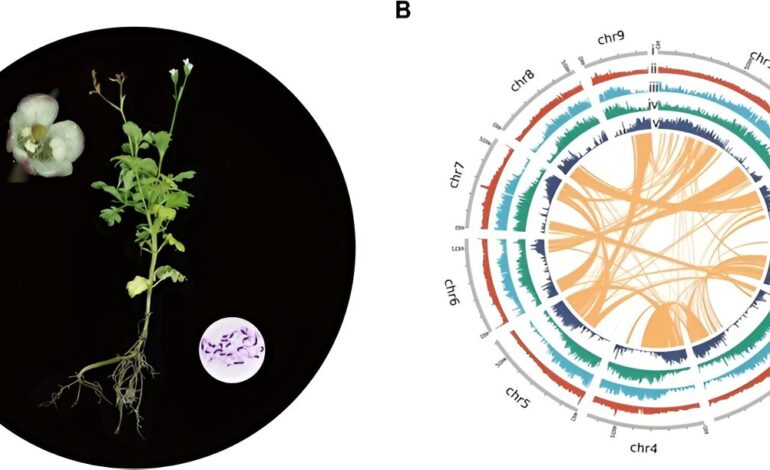
Analiza synteny oparta na porównaniu 19 pseudocząsteczek zebranych dla haplofaz Malbec. Kredyt: Badania ogrodnicze (2024). DOI: 10.1093/godz./uhae080
Genomy winorośli, znane ze swojej wysokiej heterozygotyczności, stanowią poważne wyzwanie w zakresie dokładnego składania. Tradycyjne podejścia często skupiają się na liniach prawie homozygotycznych, które nie oddają pełnej różnorodności genetycznej złożonych odmian, takich jak Malbec.
Zrozumienie klonalnej zmienności fenotypowej, która znacząco wpływa na jakość wina, zwiększa tę złożoność. Aby sprostać tym wyzwaniom, szczegółowy i dokładny montaż genomiczny jest niezbędny do zrozumienia mechanizmów genetycznych napędzających zmienność klonalną i przyczyniających się do unikalnych cech winorośli Malbec.
Zespół badaczy między innymi z Instituto de Biología Agrícola de Mendoza, Instituto de Ciencias de la Vid y del Vino i Max Planck Institute for Biology Tübingen opublikował badanie w czasopiśmie Badania ogrodnicze 14 marca 2024 r. W badaniu tym przedstawiono pierwszy montaż genomu diploidalnego winorośli Malbec, wykorzystując zaawansowane technologie sekwencjonowania w celu odróżnienia dwóch haplotypów odziedziczonych po odmianach rodzicielskich.
Badanie pozwoliło na wysokiej jakości złożenie genomu winorośli Malbec przy użyciu zaawansowanych technik sekwencjonowania długich odczytów PacBio i binningu trio. Ta metoda umożliwiła rozdzielenie i złożenie dwóch haplotypów odziedziczonych po rodzicielskich odmianach Malbec, Prunelard i Magdeleine Noire des Charentes.
Zmontowany genom ujawnił znaczące regiony polimorficzne i dostarczył szczegółowych adnotacji modelu genu dla obu haplotypów. Analiza transkryptomiczna wariantów klonalnych Malbec ujawniła różnice w ekspresji genów, szczególnie podkreślając wyższą zawartość antocyjanów w niektórych klonach. Ta zwiększona zawartość antocyjanów była związana ze zwiększoną reakcją kwasu abscysynowego, co prowadziło do nadmiernej ekspresji genów zaangażowanych w metabolizm fenylopropanoidów i reakcje na stres abiotyczny.
Odkrycia te podkreślają kluczową rolę zespołów rozpoznanych na podstawie haplotypów w zrozumieniu genetycznych podstaw zmienności klonalnej i jej wpływu na cechy istotne dla jakości wina i zdolności adaptacyjnych winorośli.
Dr Luciano Calderón, jeden z głównych badaczy, stwierdził: „Ten zestaw genomu nie tylko zwiększa naszą wiedzę na temat różnorodności genetycznej Malbec, ale także dostarcza cennego zasobu do badania mechanizmów molekularnych leżących u podstaw zmienności klonalnej. Otwiera nowe możliwości hodowli i ulepszania odmian winorośli”.
Zmontowany genom Malbec oferuje kompleksowe odniesienie do przyszłych badań genetycznych i programów hodowlanych mających na celu ulepszenie odmian winorośli. Poprzez zrozumienie genetycznych podstaw cech, takich jak skład jagód i reakcje na stres, naukowcy mogą rozwijać bardziej odporne i wysokiej jakości winorośle, co ostatecznie przyniesie korzyści branży winiarskiej i pomoże stawić czoła wyzwaniom stawianym przez zmiany klimatyczne.
Więcej informacji:
Luciano Calderón i in., Diploidalny zespół genomu odmiany winorośli Malbec umożliwia analizę uwzględniającą haplotyp różnic transkryptomicznych leżących u podstaw klonalnej zmienności fenotypowej, Badania ogrodnicze (2024). DOI: 10.1093/hr/uhae080
Dostarczone przez Chińską Akademię Nauk
Cytat:Od winorośli do wina: Odszyfrowanie różnorodności genetycznej odmiany Malbec w celu uzyskania doskonałości klonalnej (2024, 28 czerwca) pobrano 28 czerwca 2024 r. z
Niniejszy dokument podlega prawu autorskiemu. Z wyjątkiem uczciwego obrotu w celach prywatnych studiów lub badań, żadna część nie może być powielana bez pisemnej zgody. Ta zawartość jest w jedynie w celach informacyjnych.