Oświetlenie mapy genów roślin
Zmiana klimatu wystawia na próbę odporność rolników i ich upraw. Aby lepiej zrozumieć, w jaki sposób rośliny reagują na swoje dynamiczne środowisko, naukowcy pogłębiają złożone dialogi biologiczne między genami ulegającymi ekspresji w komórkach roślinnych. Mapowanie ekspresji genów ma charakter informacyjny, ale tradycyjne podejścia do przestrzennego zrozumienia ekspresji genów są ograniczone.

Tatsuya Nobori, biolog roślin w Salk Institute, bada interakcje roślina-mikrob.
Instytut Salka
W niedawnym badaniu Tatsuya Nobori, biolog roślin w Instytucie Studiów Biologicznych Salk, opracował nową technikę wizualizacji wielu genów w trójwymiarowym wewnętrznym świecie roślin.1 Opublikowane w Natura Rośliny, to ukierunkowane podejście oferuje obiecujące narzędzie do zrozumienia funkcji typów i stanów komórek oraz ich interakcji ze środowiskiem w celu identyfikacji cech korzystnych dla precyzyjnej hodowli roślin.
Naukowcy powszechnie korzystają z transkryptomiki pojedynczych komórek do sekwencjonowania poszczególnych komórek z tkanki, aby zrozumieć ich reakcje na stres. Następnie generują genetycznie zmodyfikowane linie reporterowe w celu zbadania genów docelowych zidentyfikowanych w analizie transkryptomu pojedynczych komórek. Tworzenie tych roślin reporterowych jest czasochłonne i pracochłonne, a ponadto ogranicza badaczy do badania pojedynczego genu będącego przedmiotem zainteresowania. Dodatkowo techniki obrazowania wymagają pocięcia tkanki roślinnej na cienkie plasterki, co powoduje utratę informacji przestrzennej.
„Te ograniczenia znacząco utrudniają postęp w zrozumieniu analizy przestrzennej ekspresji genów” – wyjaśnił Nobori, który kierował tymi pracami. Zmotywowało to Nobori do opracowania nowego sposobu badania genów w wysokiej rozdzielczości w 3D.
Odkrycia biologiczne, w których PHYTOMap może potencjalnie pomóc, dotyczą reakcji roślin na stres, gdy są one narażone na różne stresy środowiskowe, takie jak ocieplenie lub ujemne temperatury, a nawet ataki patogenów.
– Tatsuya Nobori, Instytut Badań Biologicznych im. Salka
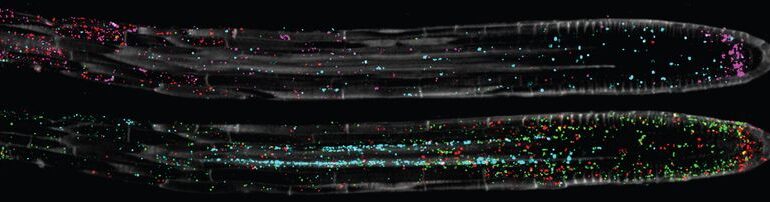
Aby zaoszczędzić czas i zbadać dziesiątki genów jednocześnie, Nobori opracował metodę zwaną ukierunkowaną obserwacją mapy ekspresji genów opartą na hybrydyzacji roślin (PHYTOMap). Oparty na technologiach hybrydyzacji i sekwencjonowania in situ, PHYTOMap jednocześnie wizualizował dziesiątki genów w przestrzeni 3D z próbką całej tkanki Arabidopsis thaliana. „Wybieram Arabidopsis jako dowód słuszności koncepcji, ponieważ był organizmem modelowym dla wielu biologów roślin i pomógł nam zrozumieć podstawowe zasady biologii roślin” – powiedział Nobori.
Celem Noboriego Arabidopsis wierzchołek korzenia, struktura krytyczna do badania wzrostu, pozyskiwania składników odżywczych i interakcji z drobnoustrojami glebowymi. Najpierw utrwalił tkankę wierzchołka korzenia całego wierzchołka, która zakotwiczyła na miejscu wszystkie docelowe cząsteczki RNA. Następnie Nobori hybrydyzował cząsteczki RNA ze sztucznymi sondami DNA zawierającymi kody kreskowe specyficzne dla genu, które amplifikował in situ i wykrywał za pomocą chemii sekwencja po hybrydyzacji. Dzięki tej technice zwizualizował określone geny, a następnie z łatwością pozbawił tkankę sond, aby zastąpić je nowym zestawem sond genów docelowych.
Aby zademonstrować możliwości multipleksowania PHYTOMap, Nobori obrał za cel 28 genów w wierzchołku korzenia. Docelowe geny obejmowały geny markerów znanych typów komórek i innych kandydatów na markery zidentyfikowanych w badaniach sekwencjonowania RNA pojedynczych komórek (scRNA-seq).2 Ponadto projekt PHYTOMap wykazywał wysoką czułość w przypadku genów o niskiej ekspresji i wychwytywał główne typy komórek i stadia rozwojowe. Kiedy Nobori skompilował obrazy, ukazało się w nich kolorowe płótno genów Arabidopsis„korzeń korzenia”.
Zobacz także „Niezbędne geny chronione przed mutacjami”
PHYTOMap wykazał dokładność porównywalną z innymi technikami obrazowania i skutecznie wykrył mRNA białka zielonej fluorescencji (GFP) w oczekiwanych typach komórek i obszarach wierzchołka korzenia Arabidopsis Model. Naukowcy wykazali przydatność PHYTOMap w badaniu przestrzennej regulacji odpowiedzi komórkowych w tkance roślinnej.
„Odkrycia biologiczne, w których PHYTOMap może potencjalnie pomóc, dotyczą reakcji roślin na stres, gdy są one narażone na różne stresy środowiskowe, takie jak ocieplenie lub ujemne temperatury, a nawet ataki patogenów” – powiedział Nobori. „Ponieważ reakcje roślin na stres są niezwykle niejednorodne, naprawdę ważne jest zrozumienie tych reakcji w rozdzielczości pojedynczej komórki z informacjami przestrzennymi”.
Praca ta reprezentuje pierwsze podejście oparte na obrazowaniu roślin, które pozwala nam wizualizować geny z rozdzielczością subkomórkową, czego brakowało w terenie.
– Stefania Giacomello, Królewski Instytut Technologii KTH i Laboratorium Nauki dla Życia
Naukowcy zajmujący się transkryptomią przestrzenną stosują dwa rodzaje podejść: metody oparte na obrazowaniu i sekwencjonowaniu. „Ta praca stanowi pierwsze podejście oparte na obrazowaniu roślin, które pozwala nam wizualizować geny z rozdzielczością subkomórkową, której brakowało w terenie” – powiedziała Stefania Giacomello, biolog przestrzenny w Kungliga Tekniska högskolan (KTH) Royal Laboratorium Instytutu Technologii i Nauki dla Życia, który nie był zaangażowany w to badanie.
W 2017 r. Giacomello opracował metodę sekwencjonowania, która uzupełnia projekt PHYTOMap jako nieukierunkowane, eksploracyjne badanie transkryptomiki roślin.3 Ta metoda eksploracyjna uchwyciła cały transkryptom, ale rozdzielczość nie była tak precyzyjna, jak w przypadku metod opartych na obrazowaniu. „To wtedy potrzebne jest ukierunkowane podejście, takie jak PHYTOMap, aby przybliżyć geny docelowe” – wyjaśnił Giacomello. Dzięki obrazowaniu w wysokiej rozdzielczości projekt PHYTOMap pobudzi wysiłki mające na celu wizualizację i uzyskanie wglądu w dynamiczne interakcje genów roślin w zmieniającym się środowisku.
Zobacz także „Siobhán Brady wykorzystuje duże zbiory danych do badania rozwoju roślin”
Ponieważ Nobori przygotowuje się teraz do założenia własnego laboratorium w Sainsbury Laboratory, planuje udoskonalić multimodalny potencjał PHYTOMap. „Widzę zrównoważenie PHYTOMap w sposób umożliwiający jednoczesne wykrywanie RNA, białek i metabolitów, aby zmaksymalizować ilość uzyskanych informacji i uzyskać lepsze zrozumienie tego, co dzieje się w interakcjach roślina-mikrob.
Bibliografia
- Nobori T. i in. Multipleksowa analiza przestrzennej ekspresji genów 3D w pojedynczych komórkach w tkance roślinnej przy użyciu PHYTOMap. Rośliny Nat. 2023;9:1026–1033.
- Shahan R. i in. Jednokomórkowa Arabidopsis Atlas korzeni ujawnia trajektorie rozwojowe mutantów typu dzikiego i mutantów tożsamości komórkowej. Komórka deweloperska. 2022;57:543–560.
- Giacomello S. i in. Przestrzennie rozwiązane profilowanie transkryptomu u modelowych gatunków roślin. Rośliny Nat. 2017;3:17061.





