Pierwszy osioł z genomem T2T melona półdzikiego
obraz:
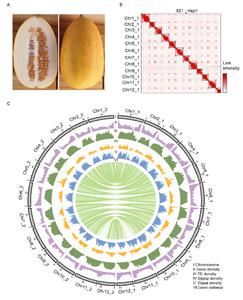
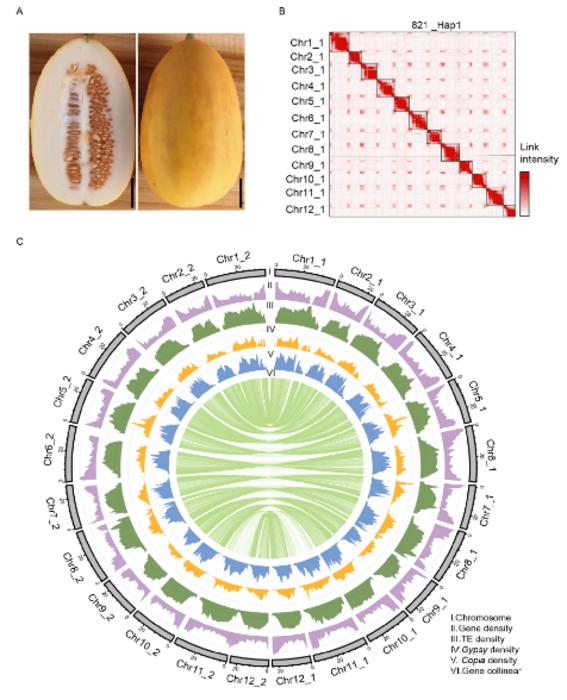
Fenotyp, mapa Hi-C i krajobraz genomowy 821
pogląd więcej
Źródło: Badania ogrodnicze
Melon (Melo Cucumis L.) to ważna roślina warzywna o bogatej historii uprawy, którą podzielono na dwa podgatunki: C. melo ssp. agrestis I C. melo ssp. melo. Poprzednie badania sugerowały, że oba podgatunki zostały udomowione niezależnie [1], co mogło wygenerować różne mechanizmy genetyczne dla tej samej cechy między dwoma podgatunkami. Co więcej, różnica w ich rozmieszczeniu geograficznym spowodowała odmienne cechy obu podgatunków, kształtując imprinting genomowy w ich genomach. Dzika plazma zarodkowa jest ważnym zasobem genetycznym w hodowli roślin uprawnych ze względu na jej dużą różnorodność genetyczną i odporność na choroby. Jednak wszystkie wcześniej zgłoszone genomy złożono na podstawie uprawianego melona[2-7], genom dzikich i półdzikich melonów nie jest jeszcze dostępny. Dlatego zgromadzenie wysokiej jakości genomu melona dzikiego/półdzikiego zapewni bezprecedensową możliwość odkrycia genów i hodowli oporności u melona.
Niedawno badanie „Genom referencyjny T2T z rozpoznanym haplotypem podkreśla zmienność strukturalną leżącą u podstaw cech agronomicznych melona’ ukazało się Badania ogrodnicze Yongyang Xu z Zespołu Genetycznej Hodowli Melonów Instytutu Badań nad Owocami w Zhengzhou Chińskiej Akademii Nauk Rolniczych współpracował z profesorem Tao Lin z Chińskiego Uniwersytetu Rolniczego.
PI 313970 to dodatek wywodzący się z C. melo ssp. agrestis odm. kwaskowaty pochodzi z Indii i charakteryzuje się wysoką odpornością na mączniaka prawdziwego i różne choroby wirusowe (ToLCNDV, CYSDV, CABYV, WmCSV i CuLCrV). Przystąpienie „821” było samozapłodnieniem z PI 313970 przez kilka pokoleń. Tutaj opisaliśmy montaż genomu T2T na poziomie chromosomu dla 821 (C. melo ssp. agrestis odm. kwaskowaty), półdziki melon z dwoma haplotypami odpowiednio ~ 373 Mb i ~ 364 Mb (ryc. 1).
Porównawcza analiza genomu odkryła znaczną liczbę wariantów strukturalnych (SV) pomiędzy melo I agrestis genomy, w tym zmienność liczby kopii zlokalizowaną w locus oporności na ToLCNDV na chromosomie 11, z którego zidentyfikowano gen-kandydat dla oporności na ToLCNDV (ryc. 2). Ponadto melon jest wyjątkowym gatunkiem modelowym do badania dojrzewania owoców, ponieważ występuje zarówno w typie klimakterycznym, jak i nieklimakterycznym. W badaniach asocjacyjnych obejmujących cały genom wykryto istotny sygnał związany z dojrzewaniem w okresie klimakterycznym i zidentyfikowano jeden gen-kandydat CM_ac12g14720.1 (CmABA2), kodujący cytoplazmatyczną dehydrogenazę/reduktazę o krótkim łańcuchu, która kontroluje biosyntezę kwasu abscysynowego. Badanie to dostarcza cennych zasobów genetycznych do przyszłych badań nad hodowlą oporności u melona.
OdnośnieodniesienieS
- Zhao, G. i in. (2019) Kompleksowa mapa zmienności genomu melona identyfikuje liczne zdarzenia udomowienia i loci wpływające na cechy agronomiczne. Nat Genet 511607-1615.
- Garcia-Mas J. i in. (2012) Genom melona (Melo Cucumis L.). PNAS 10911872-11877.
- Yang, JH i in. (2020) Genom melona w skali chromosomowej analizuje architekturę genetyczną ważnych cech agronomicznych. iNauka 23101422.
- Zhang, H. i in. (2019) Wysokiej jakości zespół genomu melona zapewnia wgląd w genetyczne podstawy poprawy cech owoców. iNauka 2216-27.
- Pichot, C. i in. (2022) Genom melona kantalupa ujawnia cechy chromatyny 3D i powiązanie strukturalne z kariotypem przodków dyniowatych. iNauka 25103696.
- Ling, J. i in. (2021) Wysokiej jakości genomy na poziomie chromosomów Cucumis metuliferus I Melo Cucumis zapewniają wgląd w ewolucję genomu Cucumis. Roślina J 107136-148.
- Castanera, R. i in. (2019) Ulepszony genom referencyjny melona za pomocą sekwencjonowania pojedynczych cząsteczek Odkrywa niedawny wybuch elementów ulegających transpozycji, które mogą mieć potencjalny wpływ na geny. Front Plant Sci 101815.
###
Autorski
Guoli Li1,2#Lingli Tang1,3,4#Yuhua He1,3Yongyang Xu1,3Abdelhafid Bendahmane5Jordi Garcia-Mas6,7Tao Lin2*Guangwei Zhao1,3,4*
Przynależności
1. Krajowe kluczowe laboratorium ds. innowacji i wykorzystania plazmy zarodkowej w uprawach ogrodniczych, Instytut Badań nad Owocami w Zhengzhou, Chińska Akademia Nauk Rolniczych, Zhengzhou, Henan, 450009, Chiny.
2. Chiński Uniwersytet Rolniczy, College of Horticulture, Pekin 100193, Chiny.
3. Narodowy Instytut Badawczy Nanfan (Sanya), Chińska Akademia Nauk Rolniczych, Sanya Hainan, 572024, Chiny
4. Centrum badawcze Zhongyuan, Chińska Akademia Nauk Rolniczych, Xinxiang 453400, Chiny.
5. Instytut Nauk o Roślinach Paris-Saclay (IPS2), INRAE, CNRS, Uniwersytet Paris-Saclay, Uniwersytet Evry, Uniwersytet Paris-Diderot, Gif sur Yvette 91192, Francja.
6. Centrum Badań nad Genomiką Rolniczą (CRAG) CSIC-IRTA-UAB-UB, Edifici CRAG, Campus UAB, 08193 Bellaterra, Barcelona, Hiszpania.
7. Institut de Recerca i Tecnologia Agroalimentàries (IRTA), Edifici CRAG, Campus UAB, 08193 Bellaterra, Barcelona, Hiszpania.
O Guangwei Zhao
Guangwei Zhao, lekarz, promotor, zajmujący się hodowlą genetyczną melona, e-mail: zhaoguangwei@caas.cn
Dziennik
Badania ogrodnicze
Metoda badań
Badania eksperymentalne
Przedmiot badań
Nie dotyczy
Tytuł artykułu
Genom referencyjny T2T z rozpoznaniem haplotypu podkreśla zmienność strukturalną leżącą u podstaw cech agronomicznych melona
Data publikacji artykułu
9 kwietnia 2023 r
Oświadczenie dotyczące COI
Autorzy deklarują, że nie mają konkurencyjnych interesów.
Zastrzeżenie: AAAS i EurekAlert! nie odpowiadają za dokładność komunikatów prasowych publikowanych w EurekAlert! przez instytucje wnoszące wkład lub za wykorzystanie jakichkolwiek informacji za pośrednictwem systemu EurekAlert.






