Struktura genetyczna i pochodzenie populacji emu w japońskich gospodarstwach wywnioskowane z genotypowania SNP na dużą skalę w oparciu o podwójnie trawione RAD-seq

Wykrywanie SNP i różnorodność genetyczna
W pięciu populacjach zwykle mapowano 3 166 876 loci na sekwencję referencyjną, przy czym 28 676 SNP wykryto z częstością ponad 50% próbek w każdej populacji. Spośród nich wykryto 5198, 22 271, 25 484, 13 469 i 6825 miejsc polimorficznych odpowiednio w TSP, OEF2016, OEF2017, FGP i KGP (tabela 1). Wskaźniki polimorficzne 3 166 876 zmapowanych odczytów wyniosły odpowiednio 0,16%, 0,70%, 0,80%, 0,43% i 0,22% dla TSP, OEF2016, OEF2017, FGP i KGP. Liczba alleli prywatnych wyniosła 69, 1260, 3259, 636 i 154 odpowiednio w TSP, OEF2016, OEF2017, FGP i KGP. Dlatego OEF charakteryzował się wyższą zmiennością genetyczną niż inne populacje, w przeciwieństwie do niższych wartości w populacjach TSP i KGP. Oczekiwane i obserwowane heterozygotyczności (Hmi/HO) TSP, OEF2016, OEF2017, FGP i KGP wykazały niskie wartości, które wynosiły odpowiednio 0,025/0,018, 0,035/0,028, 0,040/0,034, 0,039/0,029 i 0,033/0,024. Współczynniki chowu wsobnego (FJEST) TSP, OEF2016, OEF2017, FGP i KGP wynosiły odpowiednio 0,320, 0,198, 0,175, 0,237 i 0,305. Wyniki te sugerują, że wszystkie badane populacje hodowlane w Japonii mają tendencję do chowu wsobnego i że TSP jest w najgłębszym stanie, co jest zgodne z naszymi wcześniejszymi raportami5,27. Analiza rodowodu przy użyciu oprogramowania Colony2 wykazała, że w analizowanych 253 osobnikach występowały trzy pary pełnego rodzeństwa i 69 półrodzeństwa (ryc. S2). Wśród nich w TSP stwierdzono 3 rodzeństwa pełnego i 53 rodzeństwa przyrodniego, co wskazuje na postępujący chów wsobny tej populacji.
Drzewa filogenetyczne
Odległość genetyczna (FST) wśród populacji wahała się od 0,004 do 0,037 (Tabela S2). Najniższy i najwyższy FST wartości zaobserwowano odpowiednio pomiędzy OEF2016 i OEF2017 (0,004) oraz pomiędzy KGP i TSP (0,037). Drzewo filogenetyczne oparte na FST wykazało, że FGP i KGP utworzyły klad o stosunkowo dużym dystansie genetycznym między nimi, który różnił się od kladu złożonego z OEF2016, OEF2017 i TSP (ryc. 1A). Drzewo filogenetyczne, oparte na odległości genetycznej między 253 osobnikami pochodzącymi ze wszystkich badanych populacji, wykazało cztery skupienia skonstruowane przez TSP, FGP/KGP i oddzielne OEF (ryc. 1B). Na kladogramie pokazanym na ryc. 1B linie zostały wyraźniej narysowane dla wiarygodnych rozbieżności niż dla niepewnych rozbieżności. Na wczesnym etapie przewidywano, że FGP i KGP będą oddzielone od wspólnych przodków wszystkich badanych populacji japońskich hodowli emu. Kilka osobników pochodzących z OEF skupiło się w kladach różniących się od wielu innych członków i miało zamknięty związek z populacją TSP. Wyniki te sugerują, że FGP/KGP może pochodzić z innej populacji australijskiej niż reszta populacji w Japonii, a OEF składa się z dwóch rozbieżnych grup, z których jedna ma wspólnych przodków z kilkoma osobnikami z TSP.
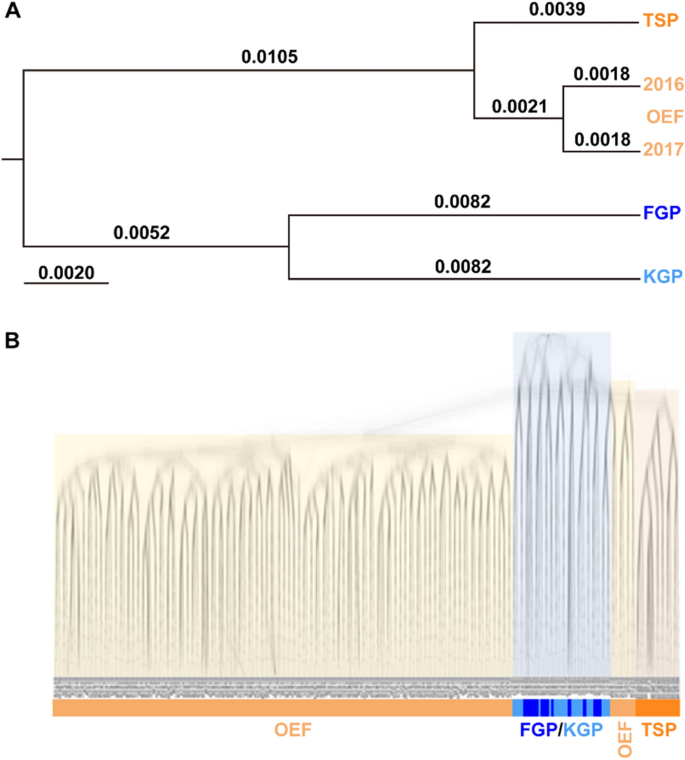
Drzewa filogenetyczne populacji japońskich emu hodowlanych na podstawie genotypów SNP. (A) Drzewo filogenetyczne wśród czterech populacji na podstawie FST obliczono na podstawie genotypów SNP. FST wartości zostały wskazane w każdym oddziale. (B) Drzewa filogenetyczne 253 osobników emu w oparciu o oprogramowanie Beast obliczone przy użyciu genotypów SNP. Węzeł żółty, niebieski i pomarańczowy (A, B) i blanszować (B) kolory oznaczały odpowiednio OEF, KGP/FGP i TSP. OEF, Ochocka Farma Emu; TSP, Park Safari Tohoku; FGP, park ogrodowy Fuji Kachoen; KGP, park ogrodowy Kakegawa Kachoen.
Analiza domieszek
Aby oszacować zmienność genetyczną w obrębie populacji i rozbieżność między populacjami, przeprowadzono analizę strukturalną przy użyciu oprogramowania ADMIXTURE. Na K= 2, struktura genetyczna pokazała, że TSP i FGP/KGP mają podobne składniki, które różnią się od tych w OEF (ryc. 2A, górny). W K= 3, chociaż TSP i FGP/KGP były przeważnie reprezentowane odpowiednio w kolorze monochromatycznym K = 2, wykazywały między sobą odrębne komponenty genetyczne (ryc. 2A, środek ). OEF ma złożoną strukturę genetyczną, obejmującą TSP i kilka składników FGP/KGP. Na K= 4, TSP było całkowicie reprezentowane przez jeden kolor (Adm1), a OEF miał wszystkie komponenty genetyczne pochodzące z TSP i FGP/KGP (Adm2), oprócz ich własnych, unikalnych komponentów (ryc. 2A, spód). Wyniki te wskazują, że TSP i FGP/KGP mają w dużej mierze różne komponenty genetyczne i że OEF powstał poprzez zmieszanie podziału przodków z TSP i kilkoma FGP/KGP.
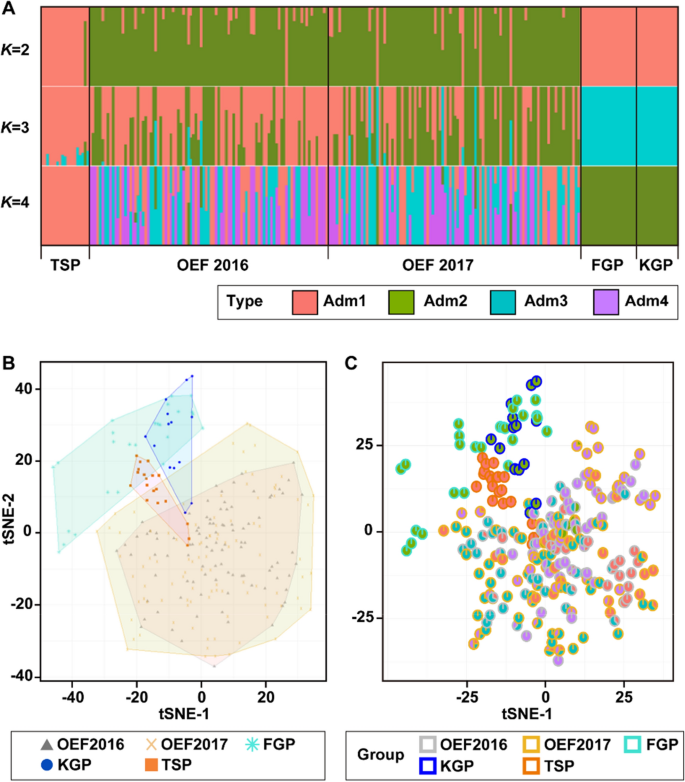
Struktura genetyczna i zależności między populacjami hodowanych japońskich emu. (A) Analiza ADMIXTURE czterech populacji emu na podstawie danych o genotypie SNP. Każde genotypowane emu jest oznaczone pojedynczą pionową linią podzieloną na Kkolory (czerwony, zielony, lodowy błękit i fioletowy), gdzie Kjest przypuszczalną liczbą przodków. (B) Wykres tSNE przedstawiający 253 osobniki emu na podstawie danych SNP. Pięć grup zostało połączonych wielokątami o różnych kolorach. Szary (trójkąty), żółty (krzyżyki), lodowoniebieski (płatki śniegu), niebieski (kółka) i pomarańczowy (kwadraty) reprezentują odpowiednio OEF2016, OEF2017, FGP, KGP i TSP. (C) Wykres kołowy tSNE przedstawiający 253 osobniki emu na podstawie danych o genotypie SNP. Każdy indywidualny punkt pokazywał wykres kołowy podzielonego klastra genetycznego za pomocą analizy domieszek (A). Pokolorowane zewnętrzne granice wykresów kołowych w kolorze szarym, żółtym, lodowoniebieskim, niebieskim i pomarańczowym wskazywały osobniki pochodzące odpowiednio z OEF2016, OEF2017, FGP, KGP i TSP. OEF, Ochocka Farma Emu; TSP, Park Safari Tohoku; FGP, park ogrodowy Fuji Kachoen; KGP, park ogrodowy Kakegawa Kachoen.
PCoA z wykresem tSNE
Wykres stochastycznego osadzania sąsiadów z rozkładem t (tSNE) oparty na genotypach SNP zgrupował populacje OEF, TSP i FGP/KGP, z wyjątkiem kilku osobników (ryc. 2B). Podobnie do wyników przedstawionych na ryc. 1, OEF był szeroko rozpowszechniony w tych zmniejszonych wymiarach, co wskazuje, że charakteryzował się różnymi zmianami genetycznymi w porównaniu z innymi populacjami. Prawie wszystkie osoby KGP pokrywały się z grupą FGP. Rozkład tSNE w grupach OEF i FGP był całkowicie oddzielony i były one bardzo odległe. Natomiast rozmieszczenie kilku osób z grup TSP i KGP pokrywało się z rozmieszczeniem osób z grupy OEF. Duża część osobników TSP była zbiorowo rozmieszczona w sąsiedztwie grupy FGP/KGP.
Następnie zintegrowaliśmy wyniki analizy domieszek i wykresu tSNE, jak pokazano na ryc. 2C. Wykresy kołowe każdego osobnika przypisanego w wyniku analizy domieszek zmapowano na wykres tSNE w celu wizualizacji struktury genetycznej i odległości genetycznej między wszystkimi próbkami (ryc. 2C). Zgodnie z powyższymi wynikami grupy FGP (ramka w kolorze lodowatoniebieskim) i KGP (ramka w kolorze niebieskim) utworzyły odrębne skupiska od grupy OEF (ramki w kolorze szarym i żółtym), a ich komponenty genetyczne składały się wyłącznie z Adm2 (ramka w kolorze zielonym kolorowy wykres kołowy), czego nie zaobserwowano w pozostałych populacjach. OEF podzielono na trzy skupienia (Adm1, Adm3 i Adm4), gdy przypuszczalna liczba przodków (K) wyniosła cztery w analizie domieszki (ryc. 2A). Informacje dotyczące skupień zintegrowane z wykresem rozproszenia tSNE uzyskane z analizy domieszek wykazały, że OEF był szeroko rozłożony od współrzędnych w przybliżeniu – 25 w tSNE-1 do 25 w tSNE-2, z wieloma skupieniami dla każdego składnika genetycznego. Populacja OEF składa się z trzech podgrup: Adm1 (przedstawiona na pomarańczowym wykresie kołowym), Adm3 (przedstawiona na niebiesko-niebieskim wykresie kołowym) i Adm4 (przedstawiona na fioletowym wykresie kołowym). Podgrupy te pochodziły od różnych przodków i wykazywały znaczny rozrzut na wykresie tSNE, co sugeruje większy stopień różnorodności genetycznej niż inne populacje.
Adm1, który był zajmowany przez osoby pochodzące z TSP, kiedy K= 4, wykazywały wyraźne rozkłady w pobliżu klastrów KGP/FGP oraz w obrębie klastra OEF na współrzędnych (− 20,0, 21,4) i (− 3,7, − 1,7) odpowiednio w wymiarze tSNE-1 i tSNE-2. W przypadku niektórych osobników z TSP w obrębie populacji śladu środowiskowego organizacji obszary te pokrywały się. Można zatem wywnioskować, że niektóre osobniki TSP mają wspólne pochodzenie przodków z populacją OEF.






