Wgląd w rozwój roślin i choroby człowieka
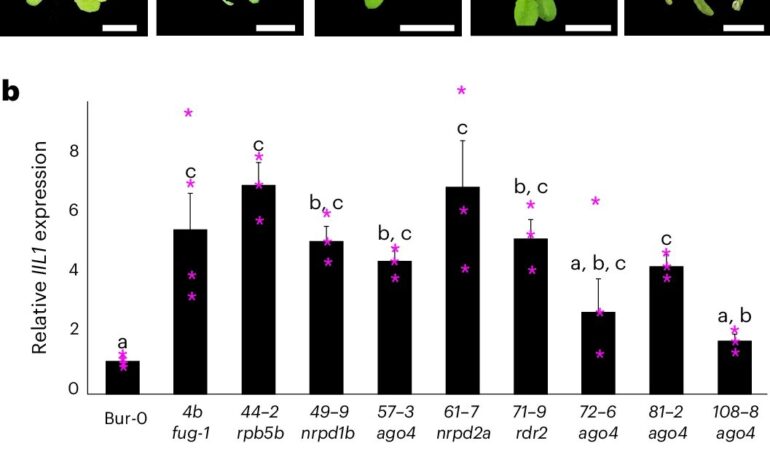
Badania przesiewowe supresora genetycznego identyfikują RdDM jako główną ścieżkę wyciszania epigenetycznego wywołanego powtarzalną ekspansją. A, Fenotypy (oznaczone przez ich oryginalne identyfikatory ekranowe) izolowanych supresorów w porównaniu z Bur-0. Liście nieregularnie uszkodzone w typie dzikim Bur-0 zaznaczono białymi strzałkami. Skala barowa 2 cm. BWzględny IIL1 poziomy ekspresji w supresorach genetycznych zidentyfikowane za pomocą badania genetycznego. Liczby reprezentują oryginalne identyfikatory przesiewowe, a poniżej pokazano odpowiednie geny zidentyfikowane po klonowaniu. Średnie poziomy ekspresji w oparciu o trzy repliki biologiczne dla każdej linii (z wyjątkiem Bur-0 i fuj1Gdzie N = odpowiednio 5 i 4). Gwiazdki oznaczają indywidualne punkty danych. Pwartości oparte są na jednokierunkowej analizie wariancji testem post hoc Tukeya, a linie z różnymi literami różnią się od siebie istotnie (P < 0,05). Słupki błędów reprezentują semC Przykład analizy SHOREmap za pomocą 44-2 identyfikuje mutację w Pol W . Allele o wysokiej częstotliwości (> 0,85) są zaznaczone na czerwono, a czerwone krzyżyki pokazują domniemane allele przyczynowe. Kredyt:Rośliny natury
(2024). DOI: 10.1038/s41477-024-01672-5 ×
zamknąć Badania przesiewowe supresora genetycznego identyfikują RdDM jako główną ścieżkę wyciszania epigenetycznego wywołanego powtarzalną ekspansją.A , Fenotypy (oznaczone przez ich oryginalne identyfikatory ekranowe) izolowanych supresorów w porównaniu z Bur-0. Liście nieregularnie uszkodzone w typie dzikim Bur-0 zaznaczono białymi strzałkami. Skala barowa 2 cm.B Względny IIL1 poziomy ekspresji w supresorach genetycznych zidentyfikowane za pomocą badania genetycznego. Liczby reprezentują oryginalne identyfikatory przesiewowe, a poniżej pokazano odpowiednie geny zidentyfikowane po klonowaniu. Średnie poziomy ekspresji w oparciu o trzy repliki biologiczne dla każdej linii (z wyjątkiem Bur-0 i fuj1 GdzieN = odpowiednio 5 i 4). Gwiazdki oznaczają indywidualne punkty danych.Pwartości oparte są na jednokierunkowej analizie wariancji testem post hoc Tukeya, a linie z różnymi literami różnią się od siebie istotnie ( P< 0,05). Słupki błędów reprezentują sem C Przykład analizy SHOREmap za pomocą 44-2identyfikuje mutację w Pol W. Allele o wysokiej częstotliwości (> 0,85) są zaznaczone na czerwono, a czerwone krzyżyki pokazują domniemane allele przyczynowe. Kredyt:
Rośliny natury (2024). DOI: 10.1038/s41477-024-01672-5Biolodzy z Uniwersytetu Monash rzucili światło na zawiłe mechanizmy molekularne odpowiedzialne za wyciszanie genów wywołane rozszerzonymi powtórzeniami w międzynarodowym badaniu opublikowanym dzisiaj w czasopiśmie
Rośliny natury
.
Zjawisko to powiązano z wieloma chorobami dziedzicznymi, w tym z ataksją Friedreicha u ludzi i powoduje zaburzenia wzrostu roślin takich jak Arabidopsis thaliana.
Celem badań było zrozumienie mechanizmu, dzięki któremu powiększone powtórzenia powodują wyciszenie epigenetyczne, co jest procedurą niezbędną do kontrolowania ekspresji genów.
Odkrycia nowych składników niezbędnych do procesu wyciszania badacze dokonali przy użyciu modelu rośliny, który wykazuje objawy wad wzrostu w wyższych temperaturach, ale nie w niższych.
Jak wynika z badania, za trzy najważniejsze czynniki uznano proteazę SUMO FUG1, czytnik histonów AL3 i białko chromodomeny LHP1.
„Białka te łączą się, tworząc niezbędny moduł wymagany do wyciszenia epigenetycznego wywołanego powtarzalną ekspansją” – powiedział główny autor badania, dr Sridevi Sureshkumar, który kieruje zespołem genetyki w podstawowej grupie badawczej w Szkole Nauk Biologicznych Uniwersytetu Monash.
„Nasze badania ujawniają kluczową rolę, jaką te białka odgrywają w organizowaniu wyciszania genów wyzwalanego przez zwiększone powtórzenia” – powiedział dr Sureshkumar.
„Świadomość tych systemów nie tylko przyczynia się do lepszego zrozumienia biologii roślin, ale także zapewnia wgląd w choroby dotykające ludzi” – powiedziała.
W trakcie badań wykorzystano nowoczesne genetyczne metody przesiewowe i testy dwuhybrydowe na drożdżach, aby ustalić, że FUG1, niescharakteryzowana proteaza SUMO, jest istotnym uczestnikiem wyciszania epigenetycznego. Po dalszej analizie wykazano, że FUG1 oddziałuje z AL3, który jest czytnikiem histonów, o którym wiadomo, że wiąże się z określonymi znacznikami histonów związanymi z efektywną ekspresją genów.
Ponadto naukowcy odkryli, że białko AL3 oddziałuje z LHP1, czyli białkiem chromodomenowym odgrywającym rolę w rozprzestrzenianiu się restrykcyjnych znaczników histonowych. Zarówno odwrócenie wyciszenia genu, jak i stłumienie objawów związanych z powtarzalną ekspansją były spowodowane utratą funkcji któregokolwiek z tych składników podczas eksperymentu.
„Odkrycia te podkreślają znaczenie modyfikatorów potranslacyjnych i czytników histonów w regulacji epigenetycznej” – powiedział dr Sureshkumar.
„Nasze badanie toruje drogę do dalszych badań nad rolą tych białek w różnych procesach biologicznych i chorobach człowieka” – stwierdziła.
„Odkrycia nie tylko przedstawiają potencjalne konsekwencje dla zdrowia ludzkiego, ale także przyczyniają się do naszego już zaawansowanego zrozumienia biologii roślin”.
Doktor Sureshkumar, która kierowała tym międzynarodowym badaniem z udziałem instytucji z Wielkiej Brytanii, Chin, Kanady, Indii i Australii, stwierdziła, że współpraca międzynarodowa pomogła im w poczynieniu postępów w różnych aspektach badania.
Dr Sureshkumar stwierdziła, że badania te mogą potencjalnie stać się drogą do opracowania nowych technik terapeutycznych ukierunkowanych na rozregulowanie epigenetyczne u osób cierpiących na choroby dziedziczne. Więcej informacji:Sridevi Sureshkumar i wsp., proteaza SUMO FUG1, czytnik histonów AL3 i białko chromodomeny LHP1 są integralną częścią wyciszania genów wywołanego ekspansją powtórzeń u Arabidopsis thaliana,
Rośliny natury
(2024). DOI: 10.1038/s41477-024-01672-5
Informacje o czasopiśmie:Rośliny natury






