Wpływ hiperurykemii na ryzyko PChN wykraczający poza predyspozycje genetyczne w badaniu kohortowym opartym na populacji
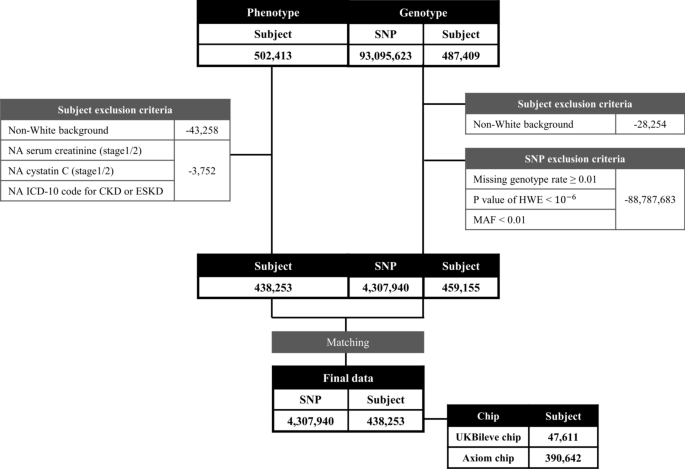
Populacje badawcze i pozyskiwanie danych
Wykorzystaliśmy dane kohortowe z prospektywnego badania United Kingdom Biobank (UKB) (https://www.ukbiobank.ac.uk)23. UKB to baza danych na dużą skalę, która obejmuje dane 502 413 osób w wieku od 40 do 69 lat. W przypadku zestawu danych fenotypowych wykluczono osoby o innym niż białe pochodzeniu, bez danych dotyczących kreatyniny w surowicy, cystatyny C, bez danych dotyczących przewlekłej choroby nerek lub przewlekłej choroby nerek na podstawie kodu ICD-10. Skupiliśmy się na białych osobach niebędących Latynosami, które stanowiły 94% kohorty UKB, co dało próbkę 438 253 osób do naszej ostatecznej analizy. W przypadku analizy wrażliwości z zestawem danych podłużnych zebraliśmy powtórzone pomiary daty uczestnictwa w ośrodku oceny, które obejmują fenotypy związane z nerkami, takie jak kreatynina w surowicy, cystatyna C, kod ICD-10 dla przewlekłej choroby nerek lub przewlekłej choroby nerek.
Definicja wyników klinicznych i narażeń
Przewlekłą chorobę nerek uznano za główny wynik, a zdefiniowaliśmy ją jako obecność jednego lub więcej z następujących stanów w kohorcie wyjściowej lub w drugim stadium w bazie danych UKB: 1) szacowany współczynnik filtracji kłębuszkowej (eGFR) < 60 ml/min/1,73 m2 na podstawie kreatyniny (pole 30 700) lub cystatyny C (pole 30 720) na podstawie równania CKD Epidemiology Collaboration (CKD-EPI); 2) kodu diagnostycznego dla stadium 3–5 CKD (N18.3–N18.5) lub schyłkowej niewydolności nerek (ESKD) (N18.6, Z94.0) na podstawie kodu Międzynarodowej Klasyfikacji Chorób, 10. rewizja (ICD-10) (pole 41 270); lub 3) zapisu rozpoznania ESKD (pole 42 026 lub 42 027).
Jako główny parametr ekspozycji przyjęto kwas moczowy jako zmienną ciągłą i kategoryjną, definiując hiperurykemię jako poziom kwasu moczowego > 420 μmol/l u mężczyzn i > 360 μmol/l u kobiet.
Genotypowanie, kontrola jakości i imputacja
Spośród 502 413 osób w badaniu UKB, 50 000 zostało genotypowanych przy użyciu UK Biobank Lung Exome Variant Evaluation (UK BiLEVE) Axiom Array firmy Affymetrix, a pozostałych osób genotypowano przy użyciu UK Biobank Axiom Array. Wstępna faza została przeprowadzona przy użyciu SHAPEIT324 z 1000 genomów fazy 3,25 a imputację nietypowanych pojedynczych polimorfizmów nukleotydowych (SNP) wykonano przy użyciu IMPUTE4 (https://jmarchini.org/software/)26 wykorzystując UK10K, 1000 Genome faza 3 i panel HRC,27 co dało 93 095 623 autosomalnych SNP. Przypisany zestaw danych został pobrany i przeprowadzono kontrolę jakości. SNP zostały odfiltrowane, jeśli wskaźniki brakujących genotypów były ≥ 0,01, równowaga Hardy’ego–Weinberga P wartości były < 10−6lub częstości występowania alleli drugorzędnych (MAF) były < 0,01. Badani zostali wykluczeni, jeśli nie byli rasy białej. Szczegółowa procedura jest zilustrowana na rys. 1. Całe zarządzanie danymi zostało przeprowadzone przy użyciu PLINK,28 PLINK2,29 GTCTA,30 i ONETOOL31.
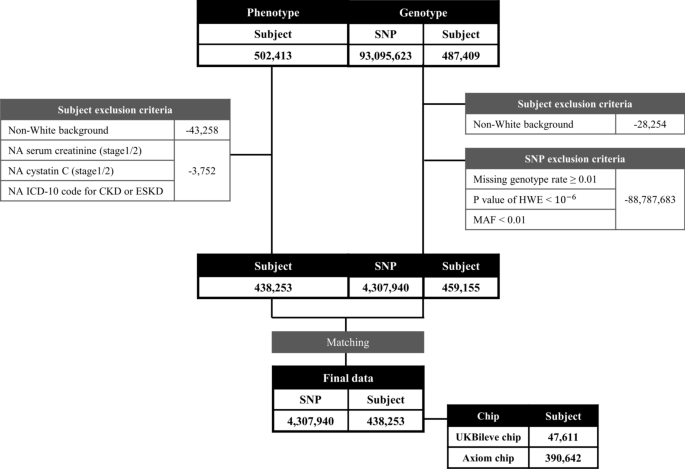
Schemat blokowy badania opartego na danych fenotypu klinicznego i genotypu.
Analizy GWAS i dziedziczności
Oszacowaliśmy dziedziczność SNP i korelacje genetyczne, wykorzystując regresję punktacji nierównowagi sprzężeń (LD)32Regresja wyniku LD wymaga podsumowujących statystyk z badań GWAS, a badania GWAS przeprowadzono na zbiorze danych UKB, stosując regresje logistyczne dla PChN i hiperurykemii po dostosowaniu do wpływu wieku wyjściowego, płci i dziesięciu głównych składowych (PC) odpowiadających 10 największym wartościom własnym.
Obliczanie PRS i jego ocena
Wyprowadzenie PRS wymaga podsumowujących statystyk GWAS, a my wzięliśmy pod uwagę następujące podsumowujące dane GWAS, które nie obejmowały zestawu danych UKB wśród opublikowanych dokumentów CKD Gen Consortium (https://ckdgen.imbi.uni-freiburg.de/)33. Wyniki z SNP z MAF ≥ 0,005 wykorzystano do PRS. PRS obliczono za pomocą grupowania + progowania (CT),34 LDpred z modelami infinitezymalnymi, siatkowymi i automatycznymi,35,36 lassosum,37 i ciągły skurcz PRS (CS)38W przypadku CT ustawiamy zbrylanie na P= 10–5 i przycinanie R2 = 0,2. W przypadku modelu siatki LDpred proporcje wariantów przyczynowych \(\rho\) zostały ustawione na 0,03, 0,01, 0,3, 0,1 i 1. Podpopulacja europejska fazy 3 1000 genomów została użyta jako panel odniesienia nierównowagi sprzężeń (LD). W przypadku innych hiperparametrów użyliśmy wartości domyślnych. Aby zbadać częstość występowania CKD jako wzrost PRS, obliczone PRS dla CKD zostały skategoryzowane w decyle. Ponadto, w przypadku regresji logistycznej, ponownie zdefiniowaliśmy grupy PRS jako zakresy tercylowe, a 2. zakres tercylowy został uznany za odniesienie w modelu regresji logistycznej do oceny wpływu PRS na częstość występowania CKD.
Aby zweryfikować wydajność PRS, podzieliliśmy UKB na dwa zestawy danych białej grupy tła, aby zweryfikować i przetestować dane. Do weryfikacji i testowania wykorzystaliśmy 47 611 osób genotypowanych przy użyciu chipa UK BiLEVE array i 390 642 osób genotypowanych przy użyciu chipa Axiom. Dane walidacyjne wykorzystano do dopasowania regresji logistycznej PRS w CKD po dostosowaniu do wieku wyjściowego, płci, PRS i PC 1–10 w wyniku CKD. Kryterium informacyjne Akaike (AIC) wykorzystano do wyboru najlepszej metody PRS.
Znaczenie PRS oceniano, stosując regresję logistyczną do danych testowych. Uwzględniliśmy wiek wyjściowy, płeć, eGFR, wskaźnik masy ciała (BMI), status palacza, aktywność fizyczną, choroby współistniejące (cukrzyca typu 2 [T2DM]nadciśnienie [HTN]choroby układu krążenia [CVD]), hemoglobina, glukoza, albumina, wapń, cholesterol i PC 1–10 jako współzmienne. Przeprowadziliśmy również analizy przeżycia przy użyciu danych testowych. Wiek w momencie wystąpienia CKD uznano za główny wynik, a te same współzmienne, co w regresji logistycznej, zostały użyte. Analizy przeżycia przeprowadzono przy użyciu pakietu survival39 (wersja 3.4.0) i pakiet ggsurvfit40(wersja 0.2.0) R (wersja 4.5.0).
Wyniki analizy PRS dla różnych modeli przedstawiono w tabeli uzupełniającej S1. Wśród SNP z wynikami GWAS od Wuttke et al.33 w UKB zaobserwowano genotypy dla 3 898 527 SNP, które wykorzystano do analiz PRS. Siatka LDpred1 (P= 0,03) uzyskał najlepszy wynik (P= 1,18 × 10–43i modelu AIC = 25 199,84) i wykorzystano go do dalszej analizy.
Analiza statystyczna
Podstawowe cechy osób z hiperurykemią i bez hiperurykemii porównano za pomocą testu t-Studenta i testu chi-kwadrat. Do oceny związku między hiperurykemią a CKD zastosowano analizę regresji logistycznej, dostosowując ją do zmiennych współistniejących, takich jak wiek, płeć, BMI, status palacza, choroby współistniejące (NT, T2DM i CVD), aktywność fizyczna, wyniki badań laboratoryjnych krwi (hemoglobina, glukoza, albumina, wapń i cholesterol), PRS i PC 1–10. Skorygowane ilorazy szans (OR) i odpowiadające im 95% przedziały ufności (CI) oszacowano dla wyników incydentalnej CKD na podstawie PRS. Aby dokładnie zbadać interakcję między hiperurykemią a PRS w przypadku CKD, włączyliśmy termin interakcji jako zmienną współistniejącą w analizie regresji logistycznej. Dodatkowo podzieliliśmy uczestników na sześć grup do analizy na podstawie zakresu tercylowego PRS i statusu hiperurykemii: PRS dla CKD 1. tercyl i hiperurykemia (−), PRS dla CKD 1. tercyl i hiperurykemia (+), PRS dla CKD 2. tercyl i hiperurykemia (−), PRS dla CKD 2. tercyl i hiperurykemia (+), PRS dla CKD 3. tercyl i hiperurykemia (−), oraz PRS dla CKD 3. tercyl i hiperurykemia (+). Grupa PRS dla CKD 1. tercyl i hiperurykemia (−) została użyta jako punkt odniesienia. Ponadto oceniliśmy wpływ hiperurykemii na rozwój CKD na podstawie płci (mężczyźni i kobiety), wieku wyjściowego (wiek < 60 i wiek ≥ 60) i cukrzycy typu 2 (grupa kontrolna i cukrzyca typu 2).
Przeprowadziliśmy analizę przeżycia Kaplana-Meiera, aby uzyskać dostęp do ryzyka rozwoju PChN wśród osób bez przewlekłej choroby nerek, które były kolejno monitorowane w bazie danych UKB. Jako zmienne niezależne wykorzystaliśmy stan hiperurykemii i stopień PRS, a jako wynik nowo zdiagnozowaną PChN. Wszystkie analizy przeprowadzono przy użyciu oprogramowania R, wersja 3.6.3 (R Foundation) i Rex. Wszystkie dwustronne wartości p były zgłaszane dwustronnie, a PZa wartość wskazującą na istotność statystyczną przyjęto < 0,0541.
Rozważania etyczne
Badanie przeprowadzono zgodnie z zasadami Deklaracji Helsińskiej i zostało ono zatwierdzone przez Institutional Review Board of Seoul National University Boramae Medical Center (IRB nr 07-2022-45). Wykorzystanie danych z UK Biobank zostało zatwierdzone przez konsorcjum UK Biobank (wniosek nr 53799). Uzyskanie świadomej zgody nie było wymagane, ponieważ badanie obejmowało anonimowe publiczne bazy danych i statystyki podsumowujące dane genetyczne.






